Los científicos explotan el abundante sistema CRISPR-Cas para editar superbacterias
Un equipo de investigación dirigido por el Dr. Aixin YAN, Profesor Asociado de la División de Investigación en Biología Molecular y Celular, Facultad de Ciencias, en colaboración con el Profesor Clínico Honorario Patrick CY WOO del Departamento de Microbiología, Facultad de Medicina Li Ka Shing, Universidad de Hong Kong (HKU), informó el desarrollo de una plataforma transferible e integradora basada en CRISPR tipo I que puede editar de manera eficiente los diversos aislados clínicos de Pseudomonas aeruginosa, una superbacteria capaz de infectar varios tejidos y órganos y una fuente importante de infecciones nosocomiales. La técnica puede acelerar la identificación de los determinantes de la resistencia en patógenos multirresistentes (MDR) y el desarrollo de nuevas estrategias anti-resistencia.
La investigación ha abierto una nueva vía para modificar genómicamente estas especies y aislados bacterianos silvestres, como los de importancia clínica y ambiental y los que forman el microbioma humano. También proporcionó un marco para explotar otros sistemas CRISPR-Cas prevalentes en genomas procarióticos y expandir los conjuntos de herramientas basados en CRISPR. La investigación ha sido publicada en la principal revista científica Nucleic Acids Research.
Fondo
El sistema CRISPR-Cas comprende el sistema inmunológico adaptativo de procariotas que desarma a los virus invasores escindiendo su ADN. Debido a su capacidad única para apuntar y modificar secuencias de ADN, CRISPR-Cas se ha aprovechado como un método de edición del genoma de próxima generación. El método se basa en el sistema CRISPR / Cas9 Clase 2 Tipo II, que revolucionó la genética y la investigación biomédica en una plétora de organismos y fue galardonado con el Premio Nobel de Química 2020. Sin embargo, los sistemas CRISPR-Cas de clase 2 representan solo ∼10 % de sistemas CRISPR-Cas codificados naturalmente en procariotas. Sus aplicaciones para editar genomas bacterianos son bastante limitadas.
Sorprendentemente, los sistemas CRISPR-Cas que pertenecen a diferentes clases y tipos se identifican continuamente y sirven como un depósito profundo para la expansión de los kits de herramientas basados en CRISPR. El sistema CRISPR-Cas más diverso y ampliamente distribuido es el sistema Tipo I que representa el 50% de todos los sistemas CRISPR-Cas identificados y tiene el potencial de expandir los juegos de herramientas basados en CRISPR con ventajas distintivas. especificidad, desvío mínimo y capaz de deleciones de grandes fragmentos. Sin embargo, el sistema CRISPR-Cas Tipo I se basa en un complejo efector multicomponente llamado Cascade para interferir con el ADN que no es fácilmente transferible a huéspedes heterólogos, lo que dificulta la aplicación generalizada de estos CRISPR naturalmente abundantes para la edición y terapia del genoma.
Principales conclusiones
Previamente, el equipo identificó un sistema IF de tipo CRISPR-Cas altamente activo en una cepa PA154197 de P. aeruginosa clínico resistente a múltiples fármacos que se aisló de un caso de infección sanguínea en el Hospital Queen Mary. Caracterizaron este sistema CRISPR-Cas y desarrollaron con éxito un método de edición del genoma aplicable en el aislado MDR basado en este sistema IF CRISPR-Cas de tipo nativo. El método permitió la identificación rápida de los determinantes de la resistencia en el aislado clínico MDR y el desarrollo de una nueva estrategia anti-resistencia (Cell Reports, 2019, 29, 1707-1717).
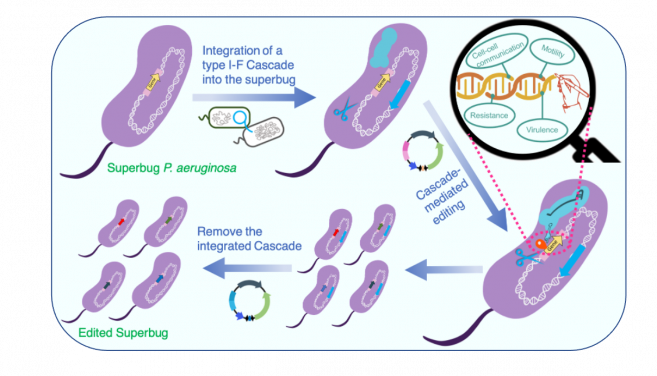
Para superar la barrera de transferencia de la cascada compleja de tipo I a huéspedes heterólogos, en este estudio, el equipo clonó todo el operón cas de tipo IF en un vector mini-CTX capaz de integración y entregó el casete a huéspedes heterólogos mediante conjugación. un enfoque de transferencia de ADN común en la naturaleza. El vector mini-CTX permitió la integración de toda la cascada en el locus genético attB conservado en el genoma de huéspedes heterólogos, lo que les permitió albergar un sistema IF CRISPR-Cas de tipo «nativo» que también puede expresarse y funcionar. . El equipo demostró que el tipo IF Cascade transferido muestra una capacidad de interferencia de ADN significativamente mayor y una mayor estabilidad de cepa que el sistema Cas9 transferible y se puede usar para la edición del genoma con eficiencia (> 80%) y simplicidad, es decir, mediante la transformación de un solo paso en un solo paso. edición de plásmido.
Además, han desarrollado un sistema transferible avanzado que comprende tanto una cascada IF altamente activa como una recombinasa para promover la aplicación del sistema en cepas con baja capacidad de recombinación homóloga, aislados silvestres de P. aeruginosa sin información de secuencia del genoma, y en otras Pseudomonas. especies. Por último, los genes similares a la cascada de IF introducidos se pueden eliminar fácilmente de los genomas del huésped mediante la eliminación inducida por la cascada de IF de grandes fragmentos de ADN, lo que da como resultado una edición del genoma sin cicatrices en las células del huésped. También se ha demostrado la aplicación del sistema transferible para la represión de genes, destacando las aplicaciones robustas y diversas del sistema transferible desarrollado IF CRISPR.
La Dra. Aixin Yan predijo que este nuevo método se extenderá para editar no solo los patógenos sino también el microbioma para promover la salud humana, dijo: “Creemos que la tecnología y las terapias basadas en CRISPR traerán nuevas esperanzas en la lucha contra las superbacterias en el futuro. «
Sobre el equipo de investigación
La investigación fue realizada por un equipo dirigido por el Dr. Aixin YAN, profesor asociado de la Facultad de Ciencias Biológicas y División de Investigación de Biología Molecular y Celular, HKU. El Dr. Zeling XU (fue becario postdoctoral en la División de Investigación de la Facultad de Ciencias Biológicas y Biología Molecular y Celular, HKU, actualmente afiliado a la Universidad Agrícola del Sur de China) es el primer autor. Otros científicos que contribuyen a la investigación son el Prof. Patrick CY WOO (Departamento de Microbiología, Facultad de Medicina Li Ka Shing, HKU), el Dr. Huiluo CAO (Departamento de Microbiología, Facultad de Medicina Li Ka Shing, HKU); Dr. Meiru SI (Universidad Normal de Qufu) y estudiantes de posgrado Sra. Yanran LI y Sr. Guangming ZHANG (Facultad de Ciencias Biológicas y División de Investigación de Biología Molecular y Celular, HKU).
El trabajo fue apoyado por el Consejo de Subvenciones de Investigación de Hong Kong (CRF y GRF), el Fondo de Investigación Médica y de Salud y el Fondo Semilla para la Investigación Básica.